if (!require('dplyr')) install.packages('dplyr'); library('dplyr')
if (!require("DT")) install.packages("DT"); library("DT")
if (!require("ggplot2")) install.packages("ggplot2"); library("ggplot2")
if (!require("here")) install.packages("here"); library("here")
if (!require("janitor")) install.packages("janitor"); library("janitor")
if (!require("purrr")) install.packages("purrr"); library("purrr")
if (!require('readr')) install.packages('readr'); library('readr')
if (!require("readxl")) install.packages("readxl"); library("readxl")
if (!require("tidyr")) install.packages("tidyr"); library("tidyr")
if (!require("waldo")) install.packages("waldo"); library("waldo")5 Combinar datos
Combinar distintos data frames es una tarea muy común cuando preparamos datos. En ocasiones trabajaremos con distintos archivos que tendremos que combinar, y otras veces, separaremos nuestra base original en distintos data frames, para realizar procesamientos diferenciados, y más adelante volver a combinar los data frames en una base final.
Paquetes para este capítulo
5.1 Bind rows or columns
El método más sencillo. Simplemente unimos las filas o columnas de los data frames.
Primero importamos dos DFs:
Con bind_rows() podemos añadir las filas de DF2 a DF1:
DF1 |> bind_rows(DF2)
#> # A tibble: 800 × 9
#> Sex Priming trialN Block Adjective Valence Answer Arrow rT
#> <chr> <chr> <dbl> <chr> <chr> <chr> <chr> <chr> <dbl>
#> 1 male Collective 1 we ofensivo negative yes left 623
#> 2 male Collective 2 we resentido negative no right 1235
#> 3 male Collective 3 we ego�sta negative yes left 335
#> 4 male Collective 4 we indiscreto negative yes left 355
#> 5 male Collective 5 we sumiso negative yes left 618
#> 6 male Collective 6 we agradable positive yes left 328
#> # ℹ 794 more rowsCon bind_cols() añadimos las columnas de DF2 a DF1. bind_cols() renombra automáticamente los nombres de las columnas para que no haya coincidencias:
DF1 |> bind_cols(DF2)
#> # A tibble: 400 × 18
#> Sex...1 Priming...2 trialN...3 Block...4 Adjective...5 Valence...6
#> <chr> <chr> <dbl> <chr> <chr> <chr>
#> 1 male Collective 1 we ofensivo negative
#> 2 male Collective 2 we resentido negative
#> 3 male Collective 3 we ego�sta negative
#> 4 male Collective 4 we indiscreto negative
#> 5 male Collective 5 we sumiso negative
#> 6 male Collective 6 we agradable positive
#> # ℹ 394 more rows
#> # ℹ 12 more variables: Answer...7 <chr>, Arrow...8 <chr>, rT...9 <dbl>, …5.2 Joins
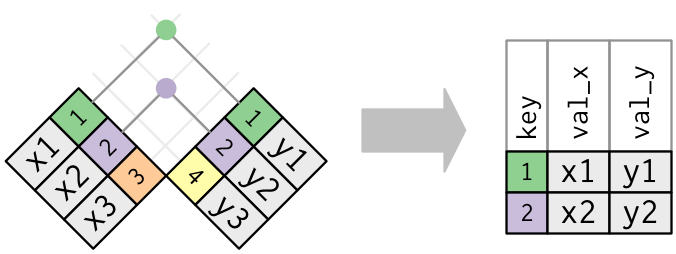
El paquete {dplyr} tiene funciones que permiten trabajar combinando, filtrando, etc. distintos data frames. Podéis ver más detalle y algunas ilustraciones fantásticas (como la de abajo; inner_join()) en el capítulo relational data de r4ds.
En https://github.com/gadenbuie/tidyexplain se pueden ver animaciones mostrando estas operaciones.
Estas operaciones tendrán la forma: DF_x |> WHATEVER_join(DF_y)
-
Mutating joins:
- inner_join(): preserva pares de observaciones de
DF_xy deDF_ycon claves iguales
- left_join(): preserva las observaciones de
DF_x, añadiendo las deDF_ycon claves iguales
- right_join(): preserva las observaciones de
DF_y, añadiendo las deDF_xcon claves iguales
- full_join(): preserva todas las observaciones de
DF_xyDF_y, alineándolas cuando tengan claves iguales
- inner_join(): preserva pares de observaciones de
-
Filtering joins:
- semi_join(): preserva solo aquellas observaciones de
DF_xcuyas claves aparezcan enDF_y
- anti_join(): preserva solo aquellas observaciones de
DF_xcuyas claves NO aparezcan enDF_y
- semi_join(): preserva solo aquellas observaciones de
-
Nesting joins:
- nest_join(): preserva las observaciones de
DF_x, añadiendo las deDF_ycon claves iguales
- nest_join(): preserva las observaciones de
5.2.1 Mutating joins
Importamos datos
Tenemos los siguientes data frames:
- DF_IDs: Variables demográficas de participantes
- DF_results: Resultados en variables de interés de participantes
- DF_BAD: Grupo de participantes “selectos”
5.2.1.1 Inner join
Preserva pares de observaciones de DF_x y de DF_y con claves iguales (fijaros en el mensaje que aparece en la Consola: Joining, by = "ID").
DF_inner_joined =
DF_IDs |>
inner_join(DF_results)
#nrow(DF_inner_joined)
DT::datatable(DF_inner_joined)5.2.1.2 Left join
Preserva las observaciones de DF_x, añadiendo las de DF_y con claves iguales (columnas con el mismo nombre).
DF_left_joined = DF_IDs |>
left_join(DF_results)
# Vemos el número de filas de cada data frame
# nrow(DF_left_joined)
# map(list("DF_left_joined" = DF_left_joined, "DF_IDs" = DF_IDs, "DF_results" = DF_results), nrow)
DT::datatable(DF_left_joined)Si no tenemos columnas con el mismo nombre en ambos data frames, tenemos que indicarle a la función a partir de que dos columnas queremos unir los data frames. Por ejemplo, con by = c("ID" = "Identificador") le decimos que la columna ID el primer data frame corresponde a Identificador del segundo data frame.
# Renombramos el identificador para que no coincidan
DF_results2 = DF_results |> rename(Identificador = ID)
# Si no hay variables en común, nos da un error:
# DF_left_joined = DF_IDs |>
# left_join(DF_results2)
# Error in `left_join()`:
# ! `by` must be supplied when `x` and `y` have no common variables.
# ℹ use by = character()` to perform a cross-join.
# Tenemos que indicar explicitamente que identificador del primer data frame (DF_IDs)
# coincide con que identificador del segundo data frame (DF_results2)
DF_left_joined = DF_IDs |>
left_join(DF_results2, by = c("ID" = "Identificador"))
# En las últimas versiones de dplyr, han implementado la función `join_by()` que
# permite usar una sintaxis algo más natural:
DF_left_joined2 = DF_IDs |>
left_join(DF_results2, by = join_by(ID == Identificador))
# Comparar si todo es =
# waldo::compare(DF_left_joined, DF_left_joined2)5.2.1.3 Full join
Preserva todas las observaciones de DF_x y DF_y, alineándolas cuando tengan claves iguales.
5.2.2 Filtering joins
5.2.2.1 Anti join
Preserva solo aquellas observaciones de DF_x cuyas claves NO aparezcan en DF_y.
# AVOID the people present in DF_BAD
DF_anti_joined = DF_IDs |>
anti_join(DF_BAD, by = "ID") |>
left_join(DF_results)
# CHECK
map(list("DF_anti_joined" = DF_anti_joined, "DF_IDs" = DF_IDs, "DF_BAD" = DF_BAD, "DF_results" = DF_results), nrow)
#> $DF_anti_joined
#> [1] 226
#>
#> $DF_IDs
#> [1] 235
#>
#> $DF_BAD
#> [1] 9
#>
#> $DF_results
#> [1] 234
DT::datatable(DF_anti_joined)5.2.2.2 Semi join
Preserva solo aquellas observaciones de DF_x cuyas claves aparezcan en DF_y. La diferencia con inner_join() es que NO se preservan las observaciones de DF_y.
# INCLUDE ONLY the people present in DF_BAD
DF_semi_joined = DF_IDs |>
semi_join(DF_BAD, by = "ID") |>
left_join(DF_results)
# CHECK
map(list("DF_semi_joined" = DF_semi_joined,
"DF_IDs" = DF_IDs,
"DF_BAD" = DF_BAD,
"DF_results" = DF_results),
nrow)
#> $DF_semi_joined
#> [1] 9
#>
#> $DF_IDs
#> [1] 235
#>
#> $DF_BAD
#> [1] 9
#>
#> $DF_results
#> [1] 234
DT::datatable(DF_semi_joined)Ejercicios JOINS
Con los DFs de abajo, haz las siguientes operaciones:
- Une los datos demográficos con los resultados.
- A la base resultante, quítale los sujetos descartados de
DF_BAD.
- Crea una nueva base con datos demográficos y resultados para los sujetos descartados.
- Comprueba si el promedio para
Crystallized Intelligencede los participantes descartados difiere de la de los no descartados.
group_by() |> summarise()
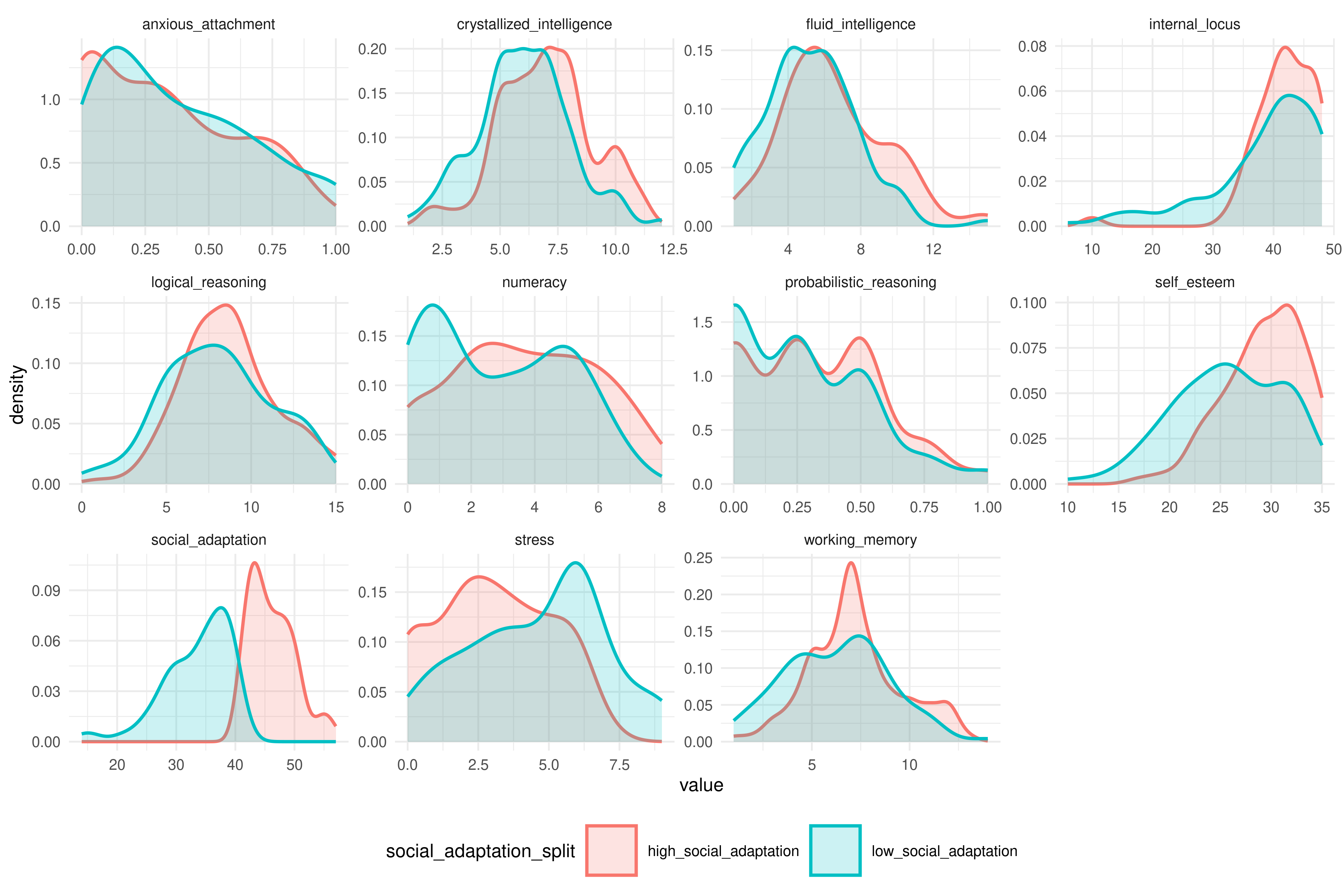
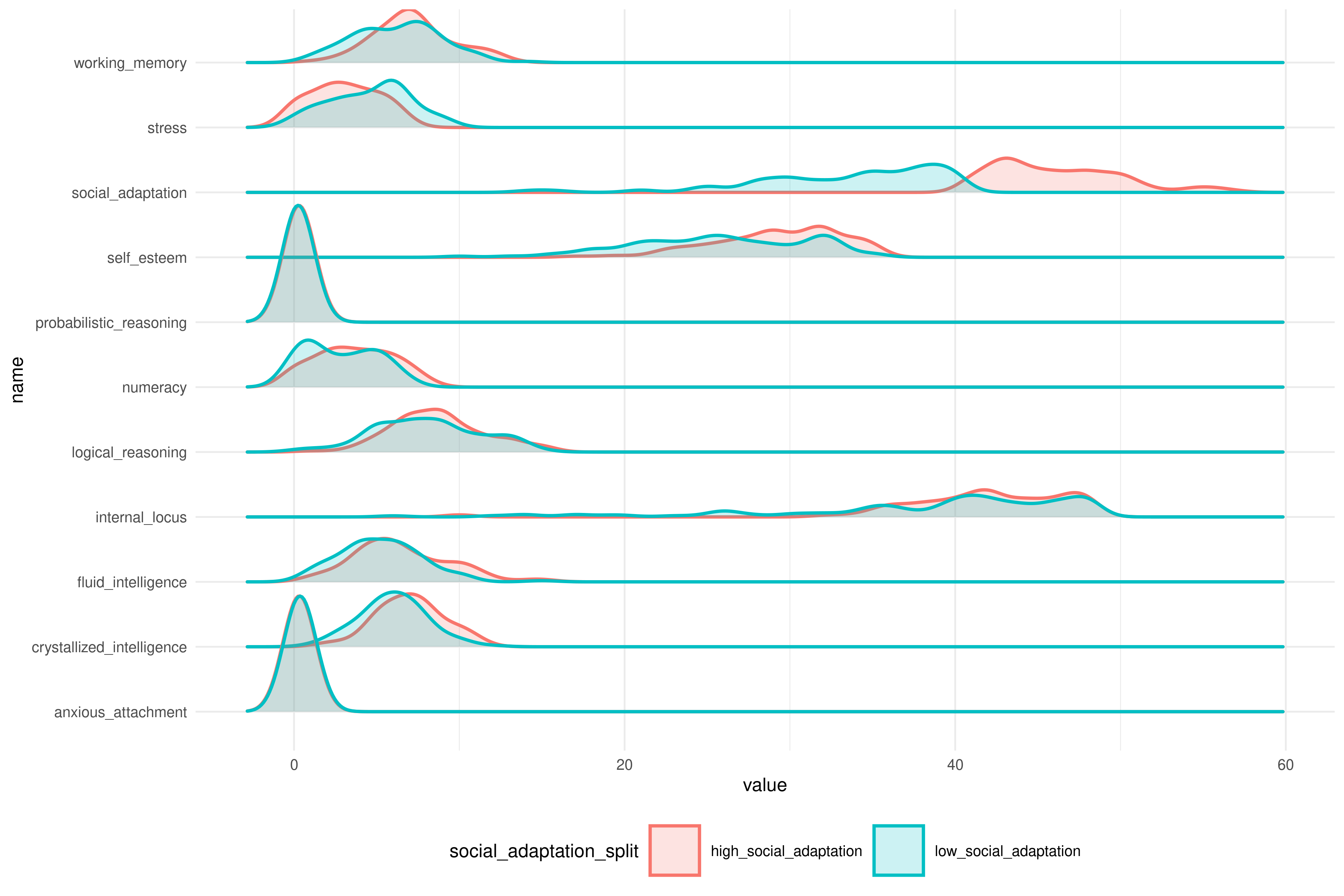
- Haz una gráfica donde se puedan ver las diferencias
- En el ejercicio 3 de verbos avanzados creaste un DF llamado
DF_splitcon la median split a partir de la variableSocial.Adaptation.
DF_wide = read_csv(
"https://raw.githubusercontent.com/gorkang/cognitive-and-socio-affective-predictors-of-social-adaptation/master/outputs/data/sa-prepared.csv"
) |>
janitor::clean_names()
median_social_adaptation = DF_wide |>
pull(social_adaptation) |>
median(., na.rm = TRUE)
DF_split = DF_wide |>
mutate(social_adaptation_split =
as.factor(
case_when(
social_adaptation >= median_social_adaptation ~ "high_social_adaptation",
social_adaptation < median_social_adaptation ~ "low_social_adaptation",
TRUE ~ NA_character_
)
)) |>
select(id, social_adaptation, social_adaptation_split) |>
drop_na(social_adaptation_split)
DF_long = DF_wide |> pivot_longer(fluid_intelligence:working_memory)Uno ese DF al DF_long que habías creado en el ejercicio 2 de la misma sección. El DF final se vera así:
- Haz un plot donde se vea la distribución para todas las variables de resultados de los dos niveles de
social_adaptation_split.
5.3 Datasets interesantes
En los siguientes repositorios podréis encontrar datasets interesantes para jugar.